Einleitung
Das in einen Inkubator eingeschlossene Hellfeldmikroskop überwacht sieben Tage lang die Differenzierung von Monozyten- zu Makrophagenzellen auf zellulärer Auflösungsebene (z. B. 2 μm). Darüber hinaus wird die Geometrie durch die Einbeziehung sehr weniger Zusatzkomponenten in ein 400-Euro-Lichtblattfluoreszenzmikroskop für volumetrische Beobachtungen eines transgenen Zebrafisches übertragen, der grün fluoreszierendes Protein (GFP) exprimiert. Damit soll ein offener Standard in der Optik etabliert werden, um die Kopplung mit verschiedenen komplementären Plattformen zu erleichtern. Indem Inhalt und umfassende Dokumentation öffentlich zugänglich sind, eignen sich die hier vorgestellten Systeme für einfache und unkomplizierte Replikationen, Modifikationen und Erweiterungen. Die wachsende Nachfrage in der biologischen Forschung nach räumlicher und zeitlicher Auflösung, Abbildungsvolumen, molekularer Spezifität und hohem Durchsatz führt zu immer komplexeren und teureren Mikroskopen [1, 2]. Neben zahlreichen bildgebenden Verfahren wurden Langzeitbeobachtungen lebender Organismen, mit minimaler Auswirkung auf deren natürliches Verhalten, zu einem wesentlichen Aspekt in der Lichtmikroskopie. Die Notwendigkeit, die Zellen in einer gut kontrollierten Umgebung zu halten, stellt zusätzliche Einschränkungen dar, die durch die Bildgebung innerhalb eines Inkubators [3, 4] oder durch die Nutzung von Inkubatoreinheiten auf Mikroskopen [5–8] angegangen werden. Der Zusammenbau, die Wartung und Verbesserung von Mikroskopen sowie die Analyse und Verifizierung der generierten Daten erfordern sehr oft einen beratenden Spezialisten, der sich mit dem jeweiligen Gerät befasst, wodurch sich die Lücke zwischen Mikroskop-Ingenieuren und Benutzern weiter vergrößert [1, 2]. Für eine Vielzahl von Bildgebungsaufgaben, wie die oben genannten, sind zwar maßgeschneiderte Lösungen kommerziell verfügbar, doch sind sie oft kostspielig, schwer zu erweitern oder zu modifizieren und selten ausreichend dokumentiert, so dass die Benutzer sie für „out-of-the-box-Aufgaben“ außerhalb ihres primären Verwendungszwecks anpassen können.
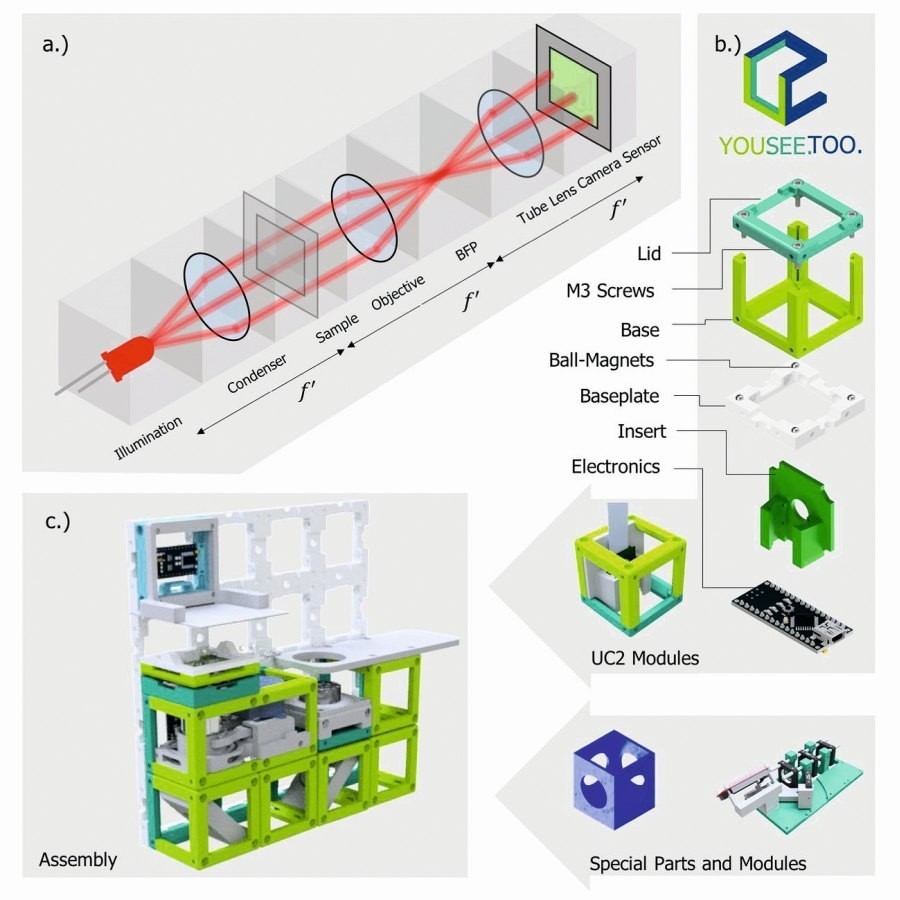 Abb. 1: a) Das 4f-System unterteilt Fourier-optische Anordnungen in funktionelle Einheiten, wobei f´ den Brennweiten entspricht. BFP entspricht der hinteren Brennebene (d.h. der Pupillenebene). b) Das Einheitselement (Würfel) dient als Grundgerüst für jede Komponente, die hineinpasst (Objektiv, Kamera, Z-Autofokussiermechanismus usw.). b) Ein magnetischer Schnapp- mechanismus verbindet die optischen Bausteine zu einem Skelett, um mechanische Stabilität und Rapid- Prototyping eines gegebenen optischen Aufbaus zu realisieren. c) Ein beispielhafter Aufbau eines Mikroskops für ein gewöhnliches Smartphone (nicht abgebildet) und ein preiswertes Objektiv als Kombination verfügbarer Module. Die Würfel passen auf das Grundplattenraster im Design-Raster von 50 × 50 mm2 (siehe Ergänzende Anmerkungen 3).
Abb. 1: a) Das 4f-System unterteilt Fourier-optische Anordnungen in funktionelle Einheiten, wobei f´ den Brennweiten entspricht. BFP entspricht der hinteren Brennebene (d.h. der Pupillenebene). b) Das Einheitselement (Würfel) dient als Grundgerüst für jede Komponente, die hineinpasst (Objektiv, Kamera, Z-Autofokussiermechanismus usw.). b) Ein magnetischer Schnapp- mechanismus verbindet die optischen Bausteine zu einem Skelett, um mechanische Stabilität und Rapid- Prototyping eines gegebenen optischen Aufbaus zu realisieren. c) Ein beispielhafter Aufbau eines Mikroskops für ein gewöhnliches Smartphone (nicht abgebildet) und ein preiswertes Objektiv als Kombination verfügbarer Module. Die Würfel passen auf das Grundplattenraster im Design-Raster von 50 × 50 mm2 (siehe Ergänzende Anmerkungen 3).
Unabhängig davon nähert sich die Wissenschaft angesichts des wachsenden Drucks, so schnell wie möglich zu publizieren, einer Reproduzierbarkeits- und Qualitätskrise [11]. Offene Forschung, bei der jeder Schritt transparent aufgezeichnet und der Öffentlichkeit uneingeschränkt zugänglich gemacht wird, kann dazu beitragen, das Vertrauen in die wissenschaftliche Literatur, das in den letzten Jahren sichtlich beeinträchtigt wurde, wiederherzustellen [12].
Moderne optische Aufbauten erreichen eine immense Komplexität, da sie eine wachsende Zahl optischer und photomechanischer Komponenten kombinieren. Sie stammen in der Regel von verschiedenen Herstellern, die sich an verschiedene Industriestandards wie die Internationale Organisation für Normung (ISO) oder die Royal Microscopy Society (RMS) halten, deren Intrakompatibilität oft nicht gewährleistet ist. Dies macht es besonders schwierig, optische Systeme maßzuschneidern oder sogar umzukonfigurieren, was handgefertigte Adapter oder unnötig lange Anbauten erfordert, die die Integrität und Stabilität der Systeme beeinträchtigen.
Was als wesentlicher Raum für Verbesserungen gesehen werden kann, ist ein offener Standard [13], der eine einfache Schnittstelle zwischen den Bestandteilen moderner Mikroskope wie Quellen, Optik, Optomechanik und Detektorkomponenten ermöglicht. Eine solche Plattform würde einfache Konstruktionen von vielseitigen Bildgebungsinstrumenten ermöglichen, die sich leicht an fast jede Bildgebungsaufgabe anpassen lassen. Der Wechsel von einem bildgebenden System zu einem anderen könnte so auf eine bloße Rekonfiguration und nicht auf ein neues Design reduziert werden. Ein solches Werkzeug wäre nicht nur für die Forschung nützlich, sondern auch in der Optik-Lehre immens hilfreich. Es würde den Aufwand für den Aufbau eines Systems erheblich reduzieren und es den Studenten ermöglichen, Systemrekonfigurationen innerhalb von Minuten aktiv durchzuführen. Eine solche praktische Erfahrung würde zu einem grundlegenden Verständnis führen und es jedem ermöglichen, die Optik als eine Spielwiese zu betrachten, auf der viele Ideen leicht erforscht werden können. Um ein solches System zu realisieren, ist ein offener Standard von entscheidender Bedeutung, da nur so ein müheloser Umbau möglich ist, ohne die Möglichkeiten allzu sehr einzuschränken. Glücklicherweise sind bereits viele große Schritte in diese Richtung gemacht worden.
Neuere Ansätze wie das Flamingo [9, 10] zielen darauf ab, die Lichtblattmikroskopie als Dienstleistung für jedermann zu etablieren und damit das Problem mangelnder Zugänglichkeit anzugehen. Eine Reihe von sehr gut dokumentierten Open-Source-Projekten wie das Lattice Light Sheet [14] oder openSPIM [15] führen zu pädagogischen Workshops und animieren damit Benutzer, zu deren Entwicklung beizutragen. Was das Hardware-Design anbelangt, so zeigen Projekte wie die „open-flexure stage“ [16], das „100-Euro-Labor“ [17], das Smartphone-basierte „Foldscope“ [18] und Open-Source-Systeme für die Einzelmolekül-Lokalisierungsmikroskopie (SMLM) [19, 20] flexible und kostengünstige Mikroskopielösungen mit hoher Leistungsfähigkeit. Generischere Ansätze wurden in Form eines opto-mechanischen Baukastens [21] und in Form einer funktionellen, kastenartigen Einheit namens μCube [22] realisiert. Mit der weit verbreiteten Verfügbarkeit von und dem einfachen Zugang zu Rapid-Prototyping-Werkzeugen wie 3D-Druck, programmierbarer Elektronik (z. B. Arduino [23]), hochwertigen Kameras in Smartphones oder Mini-Computern (Raspberry Pi [24]) ist es nun tatsächlich möglich, einen offenen Standard zu entwickeln, der für jedermann zugänglich ist und so eine weite Verbreitung, Anpassung und Erweiterung gewährleistet. Beeinträchtigungen der Bildqualität durch weniger korrigierte, preiswerte optische Komponenten oder weniger stabile mechanische Anordnungen können oft in Echtzeit durch intelligente Elektronik und Software-Algorithmen kompensiert werden. Methoden wie Autofokus-Routinen, Dekonvolution [25] oder die Wiederherstellung versteckter Informationen wie der quantitativen Phase mit einfachen LED-Arrays [26] sind aktuelle Beispiele für solche Möglichkeiten.
Mit dem UC2-Ansatz (You. See. Too) wird die Schaffung solcher offenen Standards angetrebt. Durch die Verwendung des Konzepts der übereinstimmenden Fokusebenen (Abb. 1a) ist UC2 besonders einfach zu verwenden, flexibel umkonfigurierbar und vielseitig für eine Vielzahl von Anwendungen einsetzbar. Es ist mit Open-Source-Software, offenen Designdateien und Blaupausen für eine große Vielfalt von Konfigurationen und offen zugänglicher Dokumentation ausgestattet. UC2 bietet Schülern und Studenten auf allen Ebenen eine kosten- und zeiteffiziente Möglichkeit, den Entwurf und die Anwendung einer Vielzahl komplexer optischer Setups zu erleben. Darüber hinaus ermöglicht es einer breiten Gruppe von Anwendern und Entwicklern den Zugang zur modernen Lichtmikroskopie, indem es rein handelsübliche, für den Endverbraucher erhältliche Komponenten (ergänzende Anmerkungen 1 und 8 für die Stückliste) nutzt und so kostengünstige mikroskopische Abbildungsgeräte für etwa 100 bis 400 Euro herstellt.
Das Manuskript beschreibt den gesamten Entwicklungszyklus eines Hellfeldmikroskops integriert in einen Inkubator von seiner Montage bis zur erfolgreichen Anwendung, bei der vier identische Systeme für eine parallele 168 h lange in-vitro-Bildgebungssitzung von Monozyten zu Makrophagen genutzt werden. Das Gerät wird zudem in ein Lichtblattmikroskop umgewandelt, das die ursprüngliche Hellfeldmikroskop-Baugruppe und nur einige wenige zusätzliche Komponenten nutzt. Um die Anwendbarkeit von UC2 in der biomedizinischen Forschung zu demonstrieren, werden Bildgebungsergebnisse aus einer Vielzahl biologischer Proben zur Verfügung gestellt, darunter fluoreszierende, transgene, menschliche, mikrovaskuläre Lungenendothelzellen, Drosophila melanogaster, Zebrafische, E. coli-Bakterien, die mit einer Reihe von UC2-basierten Mikroskopmodi gewonnen wurden, insbesondere dem Hellfeld-, der Weitfeldfluoreszenz-, der Bildrastermikroskopie, der Intensitätsbeugungstomographie und der strukturierten Beleuchtung.
Ergebnisse Offener Standard: Der Grundwürfel
Moderne Mikroskope mit unendlich-korrigierten Objektiven sind oft in der sogenannten 4f-Konfiguration aufgebaut (Abb. 1a), bei der die Linsen so angeordnete sind, dass die Brennebenen (f) benachbarter Elemente zusammenfallen, um die Menge der optischen Abbildungsfehler zu begrenzen, Telezentrizität zu realisieren und das Systemverhalten mittels Fourier-Optik vorherzusagen [27]. Der Name 4f ergibt sich aus der Summe der Brennweiten eines einfachen Abbildungssystems mit zwei benachbarten Linsen, die so aufgebaut sind, dass die Brennebenen aufeinanderliegen, was zu 2f pro Linse und somit zu 4f insgesamt führt. Hier ist dieses grundsätzlich modulare Prinzip in Form eines allgemeinen 3D-gedruckten Rahmens umgesetzt, in dem einzelne Module (d. h. optische Bausteine) in Würfelform (Abb. 1b und ergänzende Anmerkungen 2) so angeordnet sind, dass die Fokusebenen von Optiken in aufeinanderfolgenden Würfeln häufig zusammenfallen.
Durch die Analyse vieler verfügbarer optischer Komponenten, Abbildungssysteme und Rahmen stellte sich heraus, dass ein Design-Raster von dblock = 50 mm ein optimales Gleichgewicht zwischen Kompatibilität, Handhabung und Flexibilität für die Ermöglichung von Fourier-optischen (4f) Aufbauten zu bieten scheint. Die Aufteilung des Würfels in einen Boden und einen Deckel vereinfacht das Drucken mit Standard-Fused Deposition Modeling (FDM)-3D-Druckern und ermöglicht das einfache Einfügen von Komponenten als Plug-Ins.
Neodym-Kugelmagneten (ØMagnet = 5 mm), die gitterförmig auf einer ausziehbaren Grundplatte angebracht sind, und ferromagnetische zylindrische Bolzenschrauben (DIN 912), die in den Kanten des Würfels sitzen, ermöglichen eine stabile und präzise magnetische Befestigung. Unterschiedliche Orientierung der Grundplatten ermöglichen den Bau in drei Dimensionen. Man befand eine Vier-Punkt-Fixierung als einen guten Kompromiss zwischen der üblichen rechteckigen Anordnung der optischen Aufbauten und der mechanischen Stabilität.
Externe elektro- und optische Komponenten (z. B. Linsen, Spiegel, LEDs; siehe Abb. 1b) und bereits vorhandene Geräte (z.B. Schienensysteme von Thorlabs, Quioptics, Edmund Optics) können durch steck- und modifizierbare Einsätze leicht angepasst werden (siehe ergänzende Anmerkungen 4). Ein Modul-Entwickler-Kit (MDK, ergänzende Anmerkungen 1) mit einem generischen Referenzdesign für maßgefertigte Einsätze bietet eine einfache Schnittstelle zum Hinzufügen von Designs zur Toolbox, auch für Benutzer ohne technische Ausbildung.
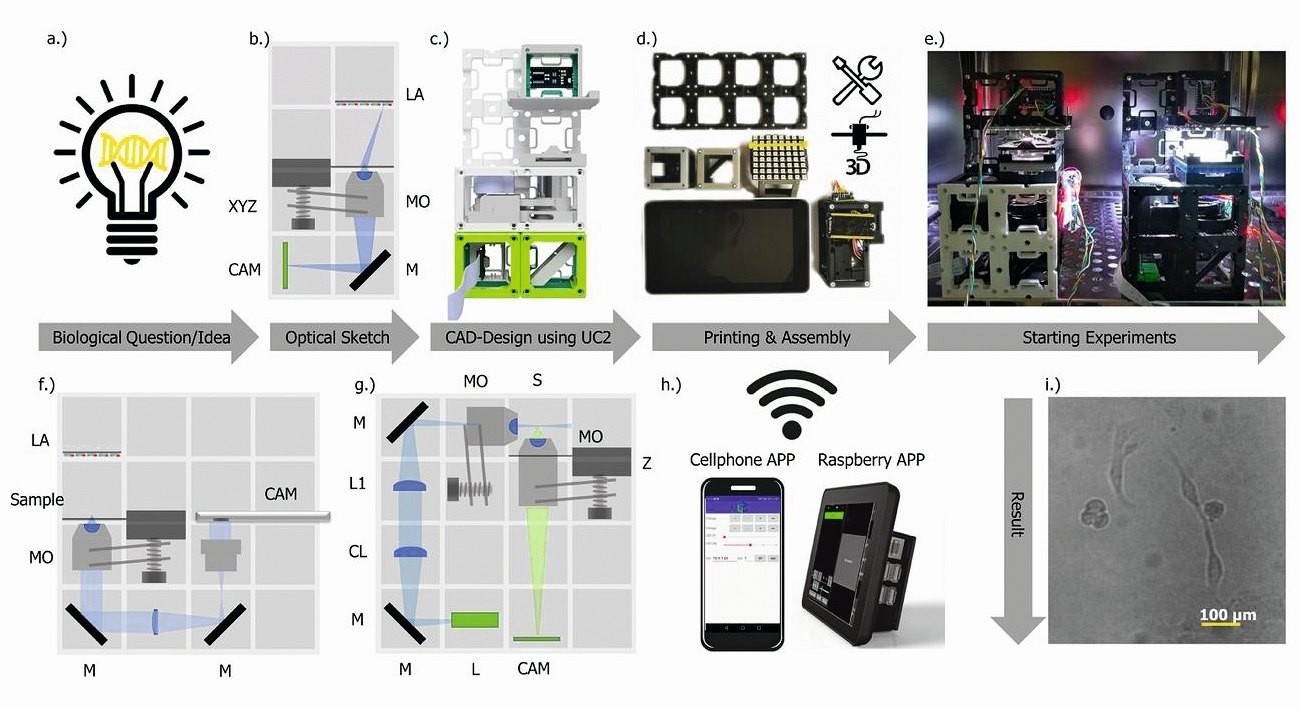 Abb. 2: Rapid Prototyping mit UC2. Üblicher Arbeitsablauf zur Erstellung einer UC2-Anwendung: (a) Beginnend mit einer biologischen Fragestellung/Idee, für die ein bildgebendes Gerät benötigt wird, das in (b) entworfen (invertiertes Inkubatormikros- kop) und in (c) mit UC2-Komponenten aus der CAD-Bibliothek umgesetzt wird. Nach dem Druck und Zusammenbau (d) wird das Gerät in seine Arbeitsumgebung (z.B. Inkubator) gestellt (e) und ist bereit, Langzeit-Bildserien von z.B. MDCK-Zellen aufzunehmen, die in (i) und im ergänzenden Video 6 visualisiert werden.
Abb. 2: Rapid Prototyping mit UC2. Üblicher Arbeitsablauf zur Erstellung einer UC2-Anwendung: (a) Beginnend mit einer biologischen Fragestellung/Idee, für die ein bildgebendes Gerät benötigt wird, das in (b) entworfen (invertiertes Inkubatormikros- kop) und in (c) mit UC2-Komponenten aus der CAD-Bibliothek umgesetzt wird. Nach dem Druck und Zusammenbau (d) wird das Gerät in seine Arbeitsumgebung (z.B. Inkubator) gestellt (e) und ist bereit, Langzeit-Bildserien von z.B. MDCK-Zellen aufzunehmen, die in (i) und im ergänzenden Video 6 visualisiert werden.
Die Fernsteuerung erfolgt über „smarte Komponenten“ (z.B. Mobiltelefon, Raspberry Pi) in (h). Die Wiederverwendung von Komponenten erlaubt den Umbau in ein Handy-Mikroskop (f) oder Lichtblattmikroskop (g) innerhalb von Minuten (siehe Ergänzungsvideo 5) und Ergänzende Hinweise 7.8). CL: Zylinderlinse, TL: Tubuslinse, L: Laser, LA: LED-Array, M: Spiegel, MO: Mikroskopobjektiv, P-CAM: Detektor (Smartphone oder Raspberry Pi), S: Probenpositioniertisch, F: Emissionsfilter, Z: Fokussiertisch
Der Aufbau zunehmend komplexerer optischer Systeme von einer einfachen Lupe bis hin zu einem voll funktionsfähigen Fluoreszenz-Lichtblattmikroskop (Abb. 2) wird durch die auf diesem Grundprinzip basierende Bibliothek an Modulen gewährleistet, welche kombiniert und in die entsprechende Reihenfolge gebracht werden (ergänzende Anmerkungen 5). Der Einsatz komplexerer kommerziell erhältlicher Komponenten (Kameras, Motoren, Videoprojekte usw.) ermöglicht den Einsatz als smartes Mikroskop und einer Fernsteuerung. Mikrocontroller erlauben eine drahtgebundene (d. h. I2C28) oder drahtlose (d. h. WiFi, IoT-basiertes Protokoll unter Verwendung des Message Queuing Telemetry Transport (MQTT) [29]) Kommunikationsschnittstelle, um Lichteinstellungen oder Fokussierungsmechanismen zu steuern (ergänzende Anmerkungen 6.1). Die Stromversorgung erfolgt über die leitenden Magnete oder Drähte mit Gleichrichtern in den Würfeln.
DIE AUTOREN
Benedict Diederich 1, 2, 3, René Lachmann 1
Swen Carlstedt 4 , Barbora Marsikova 1, 3
Haoran Wang 1 , Xavier Uwurukundo 1
Alexander S. Mosig 4 , Rainer Heintzmann 1, 2, 3
1 Leibniz Institute of Photonic Technology, Jena
2 Institute of Physical Chemistry and Abbe Center of Photonics, Jena
3 Faculty of Physics and Astronomy, Jena
4 Jena University Hospital, Institute of Biochemistry II, Jena
[1] Hell, S.W. et al.: The 2015 super-resolution microscopy roadmap, J. Phys. D: Appl. Phys. 48, 2015, 443001
[2] Weigert, M. et al.: Content-aware image restoration: pushing the limits of fluorescence microscopy, Nat. Methods 15, 2018, 1090–1097
[3] Kahle, J. et al.: Applications of a compact, easy-to-use inverted fluorescence microscope, Am. Lab. 43, 2011, 11–14
[4] Kim, J.; Henley, B.M.; Kim, C.H.; Lester, H.A.; Yang, C.: Incubator embedded cell culture imaging system (EmSight) based on Fourier ptychographic microscopy, Biomedical Optics Express 7, 2016, 3097
[5] Lukinavidius, G. et al.: Fluorogenic probes for live-cell imaging of the cytoskeleton, Nat. Methods 11, 2014, 731–733
[6] Frigault, M.M.; Lacoste, J.; Swift, J.L.; Brown, C.M.: Live-cell microscopy-tips and tools, J. Cell Sci. 122, 2009, 753–767
[7] Walzik, M.P. et al.: A portable low-cost long-term live-cell imaging platform for biomedical research and education, Biosens. Bioelectron. 64, 2014, 639–649
[8] Hernández Vera, R.; Schwan, E.; Fatsis-Kavalopoulos, N.; Kreuger, J.A.; Modular and Affordable Time-Lapse Imaging and Incubation System Based on 3D-Printed Parts, a Smartphone, and Off-The-Shelf Electronics, PLoS ONE 11(Dec), 2016, e0167583
[9] Huisken, J.; Power, R.; Bakken, T.; Li, J.; Weber, M.: Flamingo Lightsheet https://involv3d.org/concept/,2019
[10] Power, R.M.; Huisken, J.: Putting advanced microscopy in the hands of biologists. Nat. Methods 16, 2019, 1069–1073
[11] Baker, M.: 1,500 scientists lift the lid on reproducibility, https://www.nature.com/news/1-500-scientists-lift-the-lid-on-reproducibility-1.19970
[12] Fanelli, D.: Is science really facing a reproducibility crisis, and do we need it to? Proc. Natl Acad. 115, 2018, 2628–2631
[13] Faber, M.J.: Open Innovation Ansatz von Chesbrough, 21–44 https://doi.org/10.1007/978-3-83498027-4{\_} 3. https://doi.org/10.1007/978-3-8349-80274_3 (Gabler, Wiesbaden, 2009).
[14] Chen, B.-C. et al.: Lattice light-sheet microscopy: Imaging molecules to embryos at high spatiotemporal resolution, Science 346, 2014, 1257998–1257998
[15] Pitrone, P.G. et al.: OpenSPIM: An open-access light-sheet microscopy platform, Nat. Methods 10, 2013, 598–599
[16] Sharkey, J.P.; Foo, D.C.W.; Kabla, A.; Baumberg, J.J.; Bowman, R.W.: A one-piece 3D printed flexure translation stage for open-source microscopy, Rev. Sci. Instrum. 87, https://doi.org/10.1063/1.4941068, 2016
[17] Maia Chagas, A.; Prieto-Godino, L.L.; Arrenberg, A.B.; Baden, T.: The D100 lab: A 3D-printable open-source platform for fluorescence microscopy, optogenetics, and accurate temperature control during behaviour of zebrafish, Drosophila, and Caenorhabditis elegans, PLoS Biol. 15, 2017, e2002702
[18] Cybulski, J.S.; Clements, J.; Prakash, M.: Foldscope: Origami-Based Paper Microscope, PLoS ONE 9, June 2014, e98781
[19] Martens, K.J.A. et al.: Visualisation of dCas9 target search in vivo using an open-microscopy framework, Nature Communications 10, 2019, 3552
[20] Diederich, B.; Then, P.; Jügler, A.; Förster, R.; Heintzmann, R.: cellSTORMCosteffective super-resolution on a cellphone using dSTORM, PLoS ONE 14, 2019, e0209827
[21] Winters, B.J.; Shepler, D.: 3D printable optomechanical cage system with enclosure. HardwareX 3, 2018, 62–81
[22] Delmans, M.; Haseloff, J.: µ Cube: A Framework for 3D Printable Optomechanics, J. Open Hardware 2, 2018, 1–9
[23] Arduino, I.: Arduinio-open source products for electronic projects, http:// www.arduino.org/ 2019
[24] Inc., R. Raspberry Pi -Teach, Learn, and Make with Raspberry Pi. https://www.raspberrypi.org/ 2016
[25] Booth, M.J.: Adaptive optical microscopy: the ongoing quest for a perfect image. Light: Science & Applications 3, 2014, e165–e165
[26] Tian, L.; Waller, L.: Quantitative differential phase contrast imaging in an LED array microscope, Opt. Express 23, 2015, 11394
[27] Gross, H.; Singer, W.; Totzeck, M.; Gross, H.: Handbook of Optical Systems, 2006, 1–690, https://doi.org/10.1002/3527606688
[28] Semiconductors, N. UM10204 I 2 C-bus specification and user manual Rev. 64 April 2014 User manual Documen is a machine-to-machine (M2M) Internet of Things http://www.nxp.com.
[29] For the Advancement of Structured Information, S. O. MQTT https://mqtt.org/ (2019).
[30] Mantovani, A.; Sozzani, S.; Locati, M.; Allavena, P.; Sica, A.: Macrophage polarization: tumor-associated macrophages as a paradigm for polarized M2 mononuclear phagocytes, Trends Immunol. 23, 2002, 549–555
[31] Guilliams, M.; Scott, C.L.: Does niche competition determine the origin of tissue-resident macrophages? Nature Reviews Immunology, https://doi.org/10.1038/nri.2017.42, 2017
[32] Andreesen, R.; Picht, J.; Löhr, G.W.: Primary cultures of human blood-born macrophages grown on hydrophobic teflon membranes, J. Immunol. Methods 56, 1983, 295–304
[33] Jay, S.M.; Skokos, E.; Laiwalla, F.; Krady, M.M.; Kyriakides, T.R.: Foreign body giant cell formation is preceded by lamellipodia formation and can be attenuated by inhibition of Rac1 activation, Am. J. Pathol. 171, 2007, 632–640
[34] Waldo, S.W. et al.: Heterogeneity of human macrophages in culture and in atherosclerotic plaques, Am. J. Pathol. 172, 2008, 1112–1126
[35] McWhorter, F.Y.; Wang, T.; Nguyen, P.; Chung, T.; Liu, W.F. Modulation of macrophage phenotype by cell shape, Proc. Natl Acad. Sci. USA 110, 2013, 17253–17258
[36] Rosania, G.R.; Swanson, J.A.: Microtubules can modulate pseudopod activity from a distance inside macrophages, Cell Motil. Cytoskeleton. 34, 1996, 230–245
[37] Xia, Z.; Triffitt, J.T.: A review on macrophage responses to biomaterials, Biomed. Mater. 1, https://doi.org/10.1088/1748-6041/1/1/R01 2006
[38] Banterle, N.; Bui, K.H.; Lemke, E.A.; Beck, M.: Fourier ring correlation as a resolution criterion for super-resolution microscopy, J. Struct. Biol. 183, 2013, 363367
[39] Müller, C.B.; Enderlein, J.: Image Scanning Microscopy, Phys. Rev. Lett. 104, 12010, 98101
[40] Heintzmann, R.; Benedetti, P.A.: High-resolution image reconstruction in fluorescence microscopy with patterned excitation, Appl. Opt. 45, 2006, 5037–5045
[41] Diederich, B.; Wartmann, R.; Schadwinkel, H.; Heintzmann, R.: Using machine-learning to optimize phase contrast in a low-cost cellphone microscope. PLoS ONE 13, 2018, e0192937
[42] Ou, X.; Horstmeyer, R.; Zheng, G.; Yang, C.: High numerical aperture Fourier ptychography: principle, implementation and characterization, Opt. Express 23, 2015, 5473–5480
[43] Li, J. et al.: High-speed in vitro intensity diffraction tomography, Advanced Photonics 1, 2019, 1–13













